Plateforme de microscopie électronique de Tours (PFME-Tours)
Une expertise unique en microscopie électronique sur des échantillons variés : tissus, cellules, particules en suspension et macromolécules.
Plateforme de microscopie électronique de Tours (PFME-Tours)
Une expertise unique en microscopie électronique sur des échantillons variés : tissus, cellules, particules en suspension et macromolécules.
Plateforme de microscopie électronique de Tours (PFME-Tours)
La plateforme PFME-Tours offre aux équipes de recherche en biologie-santé et science des matériaux une large palette de techniques d’imagerie en microscopie, électronique à transmission, électronique à balayage et confocale (super-résolution). Les équipes utilisatrices ayant bénéficié d’une aide technique de la plateforme ont publié 75 articles dans des revues internationales de haut niveau entre 2020 et juin 2024.
La plateforme a également vocation à former les jeunes chercheurs à ces différents types de microscopies. Elle organise des formations théoriques et pratiques pour des doctorants, ainsi que des ateliers de démonstration autour des différents instruments.
La spécificité de PFME-Tours provient de son expertise en microscopie électronique, acquise pendant près de 40 années de fonctionnement en service commun. Elle est complétée par un équipement performant et un savoir-faire en microscopie photonique, notamment confocale. Depuis septembre 2024, PFME-Tours est l'une des 6 plateformes fondatrices de l'UMS61, unité mixte de service sous la tutelle de l'Université de Tours, de l'Inserm et du CHU de Tours.
Expertises et services
Microscopie électronique à transmission (MET, cryo-MET) :
- Préparation d’échantillons biologiques, y compris par des cryo-méthodes : cryo-ultramicrotomie, congélation haute pression, cryo-substitution,
- Analyse ultrastructurale de cellules et tissus,
- Immunomarquages de cellules et tissus (immunogold),
- Reconstruction 3D à partir de coupes sériées, tomographie.
Microscopie électronique à balayage (MEB, cryo-MEB) :
- Préparation d’échantillons,
- Imagerie de surface sous haute et basse tension (MEB-FEG).
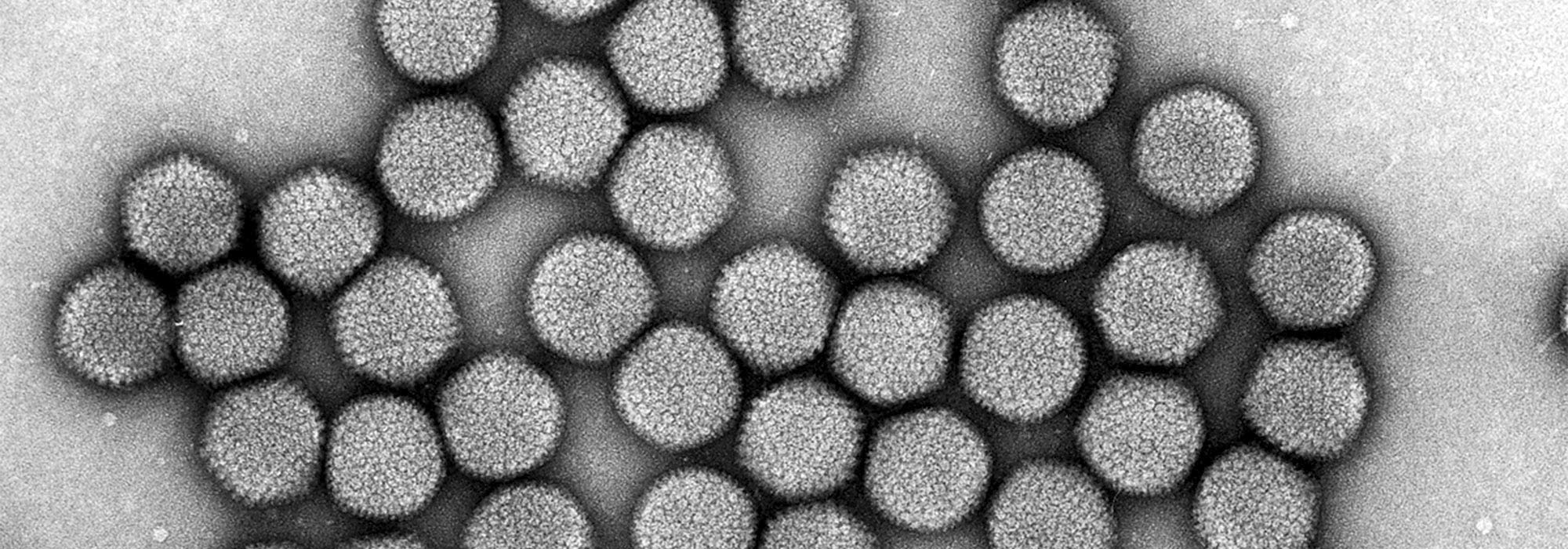
Imagerie d'objets micro ou nanométriques en MEB et MET :
- Analyse de bactéries, virus, liposomes, macromolécules, ADN et nanoparticules.
Microanalyse X par EDS en MEB et MET :
- Analyse locale et cartographie élémentaire.
Imagerie par microscopie à épifluorescence et confocale à balayage laser (CLSM) :
- Localisation d’éléments cellulaires marqués (protéines virales, facteurs de signalisation…) en 3D par microscopie confocale,
- Acquisition confocale sur cellules vivantes (time lapse),
- Reconstruction des volumes et quantification de la colocalisation de marqueurs à partir d’acquisitions en microscopie confocale,
- Imagerie par durée de vie de fluorescence (FLIM).
Moyens et équipements
Microscope électronique à transmission Jeol JEM-1400 Plus (120 kV) :
- Porte-objet cryo-Fischione Model 2550,
- Module STEM, détecteur BF/DF,
- Détecteur EDS Oxford,
- Camera CMOS Gatan OneView,
- Module de tomographie électronique et d'analyse d’images Gatan Latitude.
Microscope électronique à transmission Jeol JEM-1011 (100 kV) :
- Camera CMOS Gatan Rio 9.
Microscope électronique à balayage à effet de champ Zeiss ULTRA Plus :
- Sonde d’analyse EDS avec détecteur SDD et logiciels SAMx,
- Système de cryo-préparation et cryo-transfert Gatan Alto 2500.
Equipements pour la préparation des échantillons :
- Ultramicrotomes Leica Ultracut UCT et UC7,
- Cryo-ultramicrotome Leica EM UC7/FC7,
- Système de congélation haute pression (HPF) Leica ICE,
- Appareil de cryo-substitution Leica EM AFS,
- Station de congélation Leica EM CPC,
- Métaliseur Gatan PECS,
- Evaporateur SPI VacuPrep II,
- Station d’effluvage (glow discharge) Cordouan Elmo.
Microscope confocal à balayage laser haute résolution LEICA SP8 gSTED :
- Objectifs : 40x (NA=1.30) HC PL APO OIL, 63x (NA=1.40) HC PL APO OIL et 100x (NA=1.40) HC PL APO OIL STED White,
- Sources laser : WLL (laser blanc, jusqu’à 8 longueurs d’ondes accordables entre 470 et 670 nm), UV 405 nm et Ar 488, 514 nm (+ 458, 476 et 496 nm),
- Détecteurs : 2 PMT, 1 HyD hybride, haute sensibilité,
- Système d’acquisition en FLIM, module FRAP,
- Système à super-résolution gated-STED 2D, laser de déplétion 660 nm,
- Enceinte thermostatée avec régulation du CO2 (acquisition sur cellules vivantes),
- Station de traitement d’images avec LAS-X v2.0, Imaris v9.3 et Deconvolution SVI Huygens.
















Comment soumettre un projet ?
Pour soumettre un projet à PFME-Tours, utilisez le formulaire en ligne sur le site de la plateforme. Vous serez rapidement informé de la possibilité de prise en charge de vos analyses et dans le cas d’une réponse positive, vous recevrez un devis et un délai d’exécution.
Exemple d'utilisation
Compréhension du cycle infectieux du virus chikungunya
En collaboration avec la plateforme PFME-Tours, une équipe Inserm-CNRS du Centre de recherche Saint-Louis à Paris a démontré le rôle indispensable de la protéine cellulaire FHL1 dans la réplication du virus chikungunya, permettant de comprendre pourquoi le virus cible préférentiellement les cellules des muscles et des articulations. Les résultats montrent en particulier que chez les patients atteints de la dystrophie musculaire d'Emery-Dreifuss, les fibroblastes dermiques et les cellules musculaires sont dépourvues de FHL1 fonctionnelle, induisant une résistance à l'infection par le virus chikungunya.
Pour en savoir plus : Meertens L, et al. (2019). FHL1 is a major host factor for chikungunya virus infection. Nature, 574(7777):259-263.
Contact
PFME-Tours
Faculté de médecine
Université de Tours
10 boulevard Tonnellé
37032 Tours
Région : Centre-Val de Loire +33 (0)2 34 37 96 92
+33 (0)2 34 37 96 92
roingeard@med.univ-tours.fr
Site de la plateforme
THÉMATIQUES : Imagerie cellulaire
RESPONSABLES SCIENTIFIQUES :
Philippe Roingeard
RESPONSABLES TECHNIQUES :
Pierre-Ivan Raynal
RESPONSABLES QUALITÉ :
Christophe Hourioux
TUTELLES : CHU de Tours, Université de Tours
LABELLISATION IBiSA : 2015
MOTS CLÉS : Microscopie électronique à transmission, MET, Microscopie électronique à balayage, MEB, Tissus, Cellules, Bactéries, Particules en suspension, Virus, Macromolécules, Cryo-méthodes, Immunomicroscopie électronique
Fiche mise à jour en 2024










