Ingénierie cellulaire et analyses des protéines (ICAP)
Spectrométrie de masse et imagerie pour l’ingénierie cellulaire et l’analyse des protéines.
Ingénierie cellulaire et analyses des protéines (ICAP)
Spectrométrie de masse et imagerie pour l’ingénierie cellulaire et l’analyse des protéines.
Ingénierie cellulaire et analyses des protéines (ICAP)
ICAP est une plateforme de l’Université de Picardie Jules Verne. Localisée au sein du Centre universitaire de recherche en santé (CURS) au CHU d'Amiens, elle est spécialisée dans l’ingénierie cellulaire et l’analyse des protéines. Elle est ouverte aussi bien au secteur privé qu’au secteur public, sans aucune restriction thématique, institutionnelle ou géographique.
La plateforme met à disposition des équipements de pointe dédiés à l’identification et à l’analyse des protéines par spectrométrie de masser et imagerie cellulaire. Elle propose également un accompagnement scientifique et technique personnalisé, incluant une assistance spécialisée et si nécessaire, des formations adaptées aux utilisateurs. L’une de ses spécificités majeures réside dans sa capacité à mettre en œuvre des approches complémentaires à différentes échelles, permettant d'explorer les systèmes de manière intégrative, depuis l’organisme entier jusqu’à la molécule.
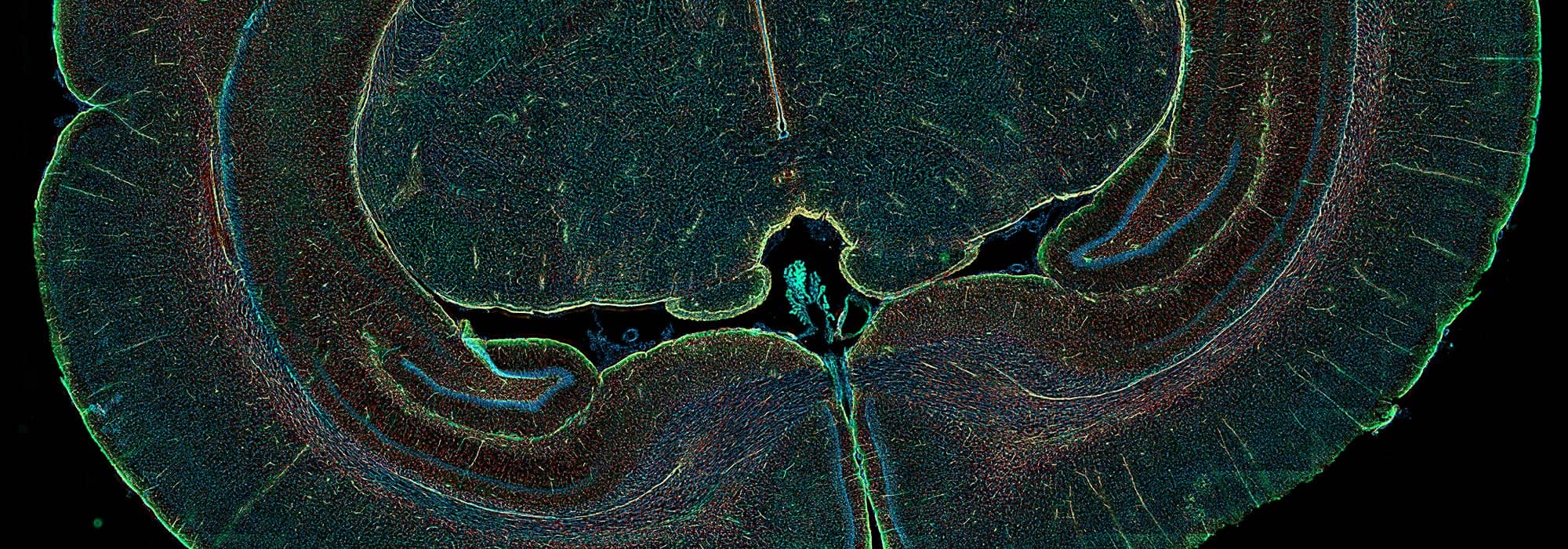
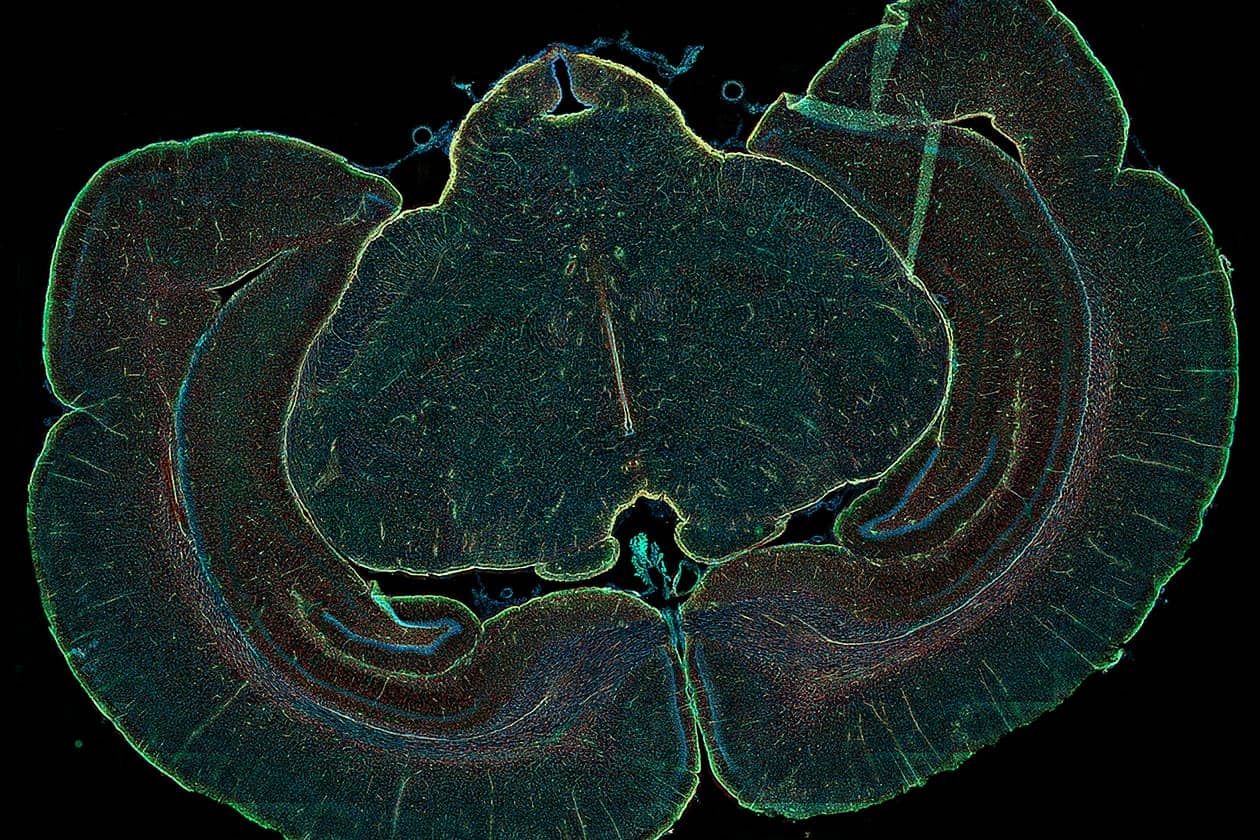
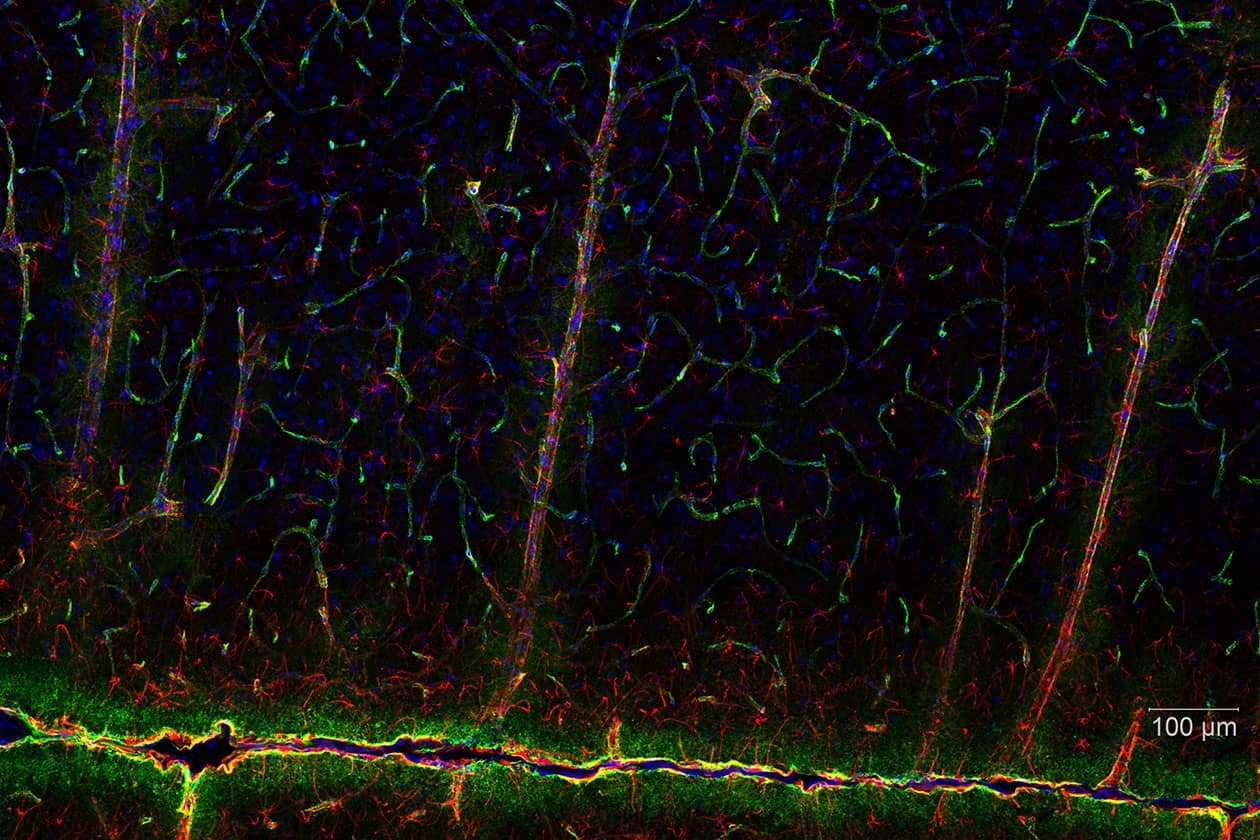
La plateforme offre un accès à un ensemble complet de techniques de bioimagerie, comprenant l’imagerie cellulaire fixe et vivante, comme la vidéomicroscopie, la microscopie confocale et la microscopie en feuillet de lumière, mais aussi la spectrométrie de masse, pour l’analyse approfondie des protéines et des petites molécules. Grâce à son expertise et à ses équipements dernier cri, elle contribue activement à des recherches innovantes et multidisciplinaires.
Expertises et services
Analyse par spectrométrie de masse :
- Identification de protéines ou peptides à partir de protéines séparées sur gel SDS, de fractions de peptides séparées par chromatographie liquide ou par approche shotgun, avec gradient court ou long selon la complexité du mélange,
- Spectrométrie de masse pour l’analyse approfondie des protéines : protéomique bottom-up et analyse quantitative par approches Silac, TMT et label-free,
- Identification de modifications post-traductionnelles : phosphorylations, glycosylations...
- Production de données métabolomiques, lipidomiques et glycomiques non ciblées par chromatographie liquide couplée à la spectrométrie de masse à haute résolution,
- Traitement de données omiques, du pré-processing à l’analyse statistique,
- Quantification absolue de protéines et de petites molécules par chromatographie liquide couplée à la spectrométrie de masse par approches MRM,
- Traitement d’échantillons : biofluides, tissus, bactéries, plantes…
Analyse par bioimagerie :
- Imagerie photonique, confocale et à feuillet de lumière avec méthodologies FRAP et FRET,
- Cytométrie en flux,
- Imagerie du petit animal par bioluminescence, rayons X et micro-tomographie,
- Animation de formations théoriques et pratiques,
- Conseil pour le design, l’acquisition et l'analyse de données,
- Sauvegarde et archivage de données.
Recherche et développement :
- Mise en place de protocoles de fractionnement et utilisation de la mobilité ionique pour l’identification de mélanges complexes,
- Mise en place d’approches protéomiques différentielles sur de larges panels d’échantillons,
- Développement d’approches d’identification et de caractérisation des vésicules extracellulaires dans différents environnements biologiques,
- Adaptation de protocoles pour la clarification de tissus, d’organes et de plantes, ou pour l’utilisation de biosensors,
- Mise en place de protocoles de reconstruction 3D en microscopie photonique,
- Préparation d’échantillons pour l’analyse chimique,
- Tri d’objets fluorescents rares et tri d’organites par cytométrie en flux,
- Imagerie et cytométrie ratiométriques.
Moyens et équipements
Spectrométrie de masse :
- Orbitrap Fusion (Thermo Fisher Scientific),
- Orbitrap Exploris 480 (Thermo Fisher Scientific),
- Xevo-TQS (Waters).
Chromatographie :
- Chaines nano-UPLC U3000 (Thermo Fisher Scientific),
- Chaine Acquity H-Class (Waters),
- Chaine Vanquish Horizon (Thermo Fisher Scientific).
Préparation échantillons :
- Robot EVO 150 (Tecan),
- Robot SPE GX241 (Gilson),
- Bioruptor Pico (Diagenode),
- BeatBox (PreOmics),
- Luminex MagPix (Merck).
Imagerie :
- Cytomètre analyseur FACS Canto II (BD Biosciences),
- Station d'imagerie Photon Imager (Biospace Lab),
- Micro-tomographe à rayons X SkyScan 1176 (Bruker),
- Système de microdissection laser PALM Microbeam (Zeiss),
- Microscope confocal LSM780 avec module Airyscan (Zeiss),
- Microscope Lightsheet Z1 (Zeiss).





Comment soumettre un projet ?
Le fonctionnement et les conditions d’utilisation de la plateforme ICAP sont détaillées sur son site internet. Pour formaliser une demande de collaboration, de prestation ou de formation, il est nécessaire de remplir le formulaire mis à votre disposition en ligne. Un rendez-vous vous sera ensuite proposé afin de mieux comprendre votre besoin et de vous exposer les différentes solutions d’accompagnement possibles. Après formation, les utilisateurs de la plateforme pourront réserver en toute autonomie des équipements sur un site dédié, avec un accès confidentiel aux données générées. En contrepartie, la contribution de la plateforme devra être mentionnée sur tous les supports de communication en lien avec le projet.
Exemple d'utilisation
Analyse protéomique quantitative pour la mise en évidence de marqueurs spécifiques des cellules souches mésenchymateuses
L’institut Faire-Face a contacté la plateforme ICAP pour mettre en évidence des marqueurs spécifiques des cellules souches mésenchymateuses (CSM) permettant leur utilisation dans un cadre thérapeutique. Ces cellules pourraient en effet constituer un outil intéressant pour l'ingénierie tissulaire et la médecine régénérative, en raison de leurs propriétés régénératives et trophiques.
Les plus connues et les plus utilisées sont les CSM de la moelle osseuse. Elles sont actuellement prélevées et développées à partir d'un large éventail de tissus adultes et périnataux. Les CSM provenant de différentes sources auraient cependant des potentiels de sécrétion et de production différents, influençant leurs effets thérapeutiques. Pour le confirmer, la plateforme ICAP a effectué une analyse protéomique quantitative basée sur la technique TMT de CSM provenant de trois sources : la gelée de Wharton (WJ), la pulpe dentaire (DP) et la moelle osseuse (BM).
L’analyse s'est concentrée sur les propriétés biologiques des CSM d'intérêt pour l'ingénierie tissulaire. Au total, il a été identifié 611 protéines humaines différentiellement exprimées. Les CSM-WJ ont montré la plus grande variation. Par rapport aux autres, elles ont produit davantage de protéines de la matrice extracellulaire (ECM) et de protéines affiliées à l'ECM, ainsi que des protéines liées aux processus de réponse inflammatoire et immunitaire. Les CSM-BM ont quant à elles exprimé davantage de protéines impliquées dans la différenciation ostéogénique, adipogénique, neuronale ou musculaire, et de protéines impliquées dans la communication paracrine. Enfin, les CSM-DP ont surexprimé des protéines impliquées dans le processus d'exocytose. Ces résultats confirment l'existence de différences entre les CSM-WJ, DP et BM et la nécessité de sélectionner l'origine des CSM en fonction de l'objectif thérapeutique recherché.
Pour en savoir plus : Naudot M. et al. (2023). TMT-based quantitative proteomics analysis reveals differentially expressed proteins between different sources of hMSCs. International Journal of Molecular Sciences, 24(17):13544.
Contact
ICAP
Centre universitaire de recherches en santé (CURS)
CHU Amiens-Picardie
80059 Amiens
Région : Hauts-de-France +33 (0)3 22 82 54 32
+33 (0)3 22 82 54 32
paulo.marcelo@u-picardie.fr
Site de la plateforme
THÉMATIQUES : Cytométrie, Imagerie cellulaire, Imagerie in vivo, radiobiologie, Métabolomique, exposome, Protéomique
RESPONSABLES SCIENTIFIQUES :
Paulo Marcelo
RESPONSABLES TECHNIQUES :
Paulo Marcelo
TUTELLES : Université de Picardie Jules Verne
LABELLISATION IBiSA : 2024
MOTS CLÉS : Bioimagerie, Cytométrie en flux, Microscopie confocale, Microscopie à feuille de lumière, Protéomique, Métabolomique, Spectrométrie de masse, Caractérisation, Quantification, Modifications post-traductionnelles, Analyse structurale, Biologie-santé, Environnement
Fiche mise à jour en 2025










